アンプリコンシーケンス解析 MiSeqデータ取得方法・解析前処理工程の一部変更のご案内
変更による解析結果への影響はございません。
アンプリコンシーケンス解析で使用しているMiSeq (Illumina, USA) につきまして、イルミナ社より機器搭載OSおよびコントロールソフトウェアのアップグレードが発表されました1)。これに伴い2021年6月7日ご依頼分より、当社アンプリコンシーケンス解析における配列データ (fastq) 取得方法および解析前処理工程が一部変更となります。なお今回の変更による解析結果への影響はございません。
前処理工程 (Cutadapt2) を経て、データ解析します。
イルミナ社のシーケンシングは一塩基伸長反応を繰り返しながら配列決定を行います。これまで当社ではDark cycleという任意の数塩基に対して配列情報を取得せずスキップする方法をプライマー配列部分に適用し、「プライマー配列を予め除去したデータ (fastq)」を取得してきました。MiSeqソフトウェアのアップグレードにより、このDark Cycleが使用できなくなります。今後は「プライマー配列を含むデータ (fastq)」を取得した後、前処理工程 (Cutadapt2)) を経て、データ解析を実施します。
変更前

変更後

-
(1)
https://jp.support.illumina.com/support-content/windows10.html
-
(2)
Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet. journal 2011;17:10-12.
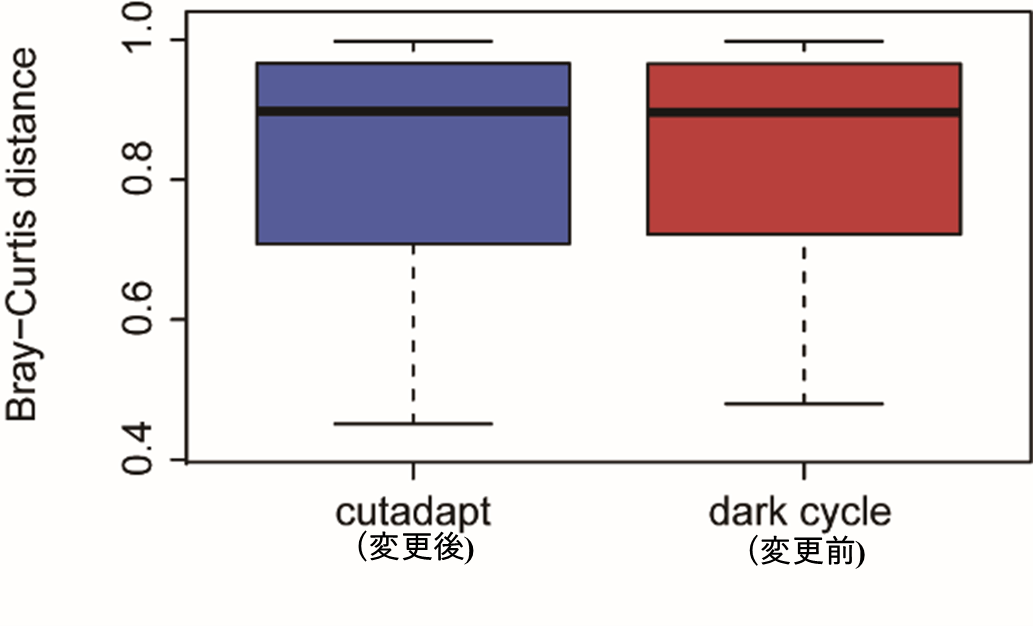
変更前後における解析結果の比較
16S rDNA (細菌・アーキア対象) のアンプリコンプール (検体数 68) に対し上記2通りの方法でデータ取得・前処理を行いRDP MultiClassifier ver.2.11.を用いて相同性解析を実施しました。
属レベルでの多様性を比較し、データ取得方法および解析前処理工程の差異は解析結果に影響しないことを確認しました。
図1. Simpson指数の相関関係
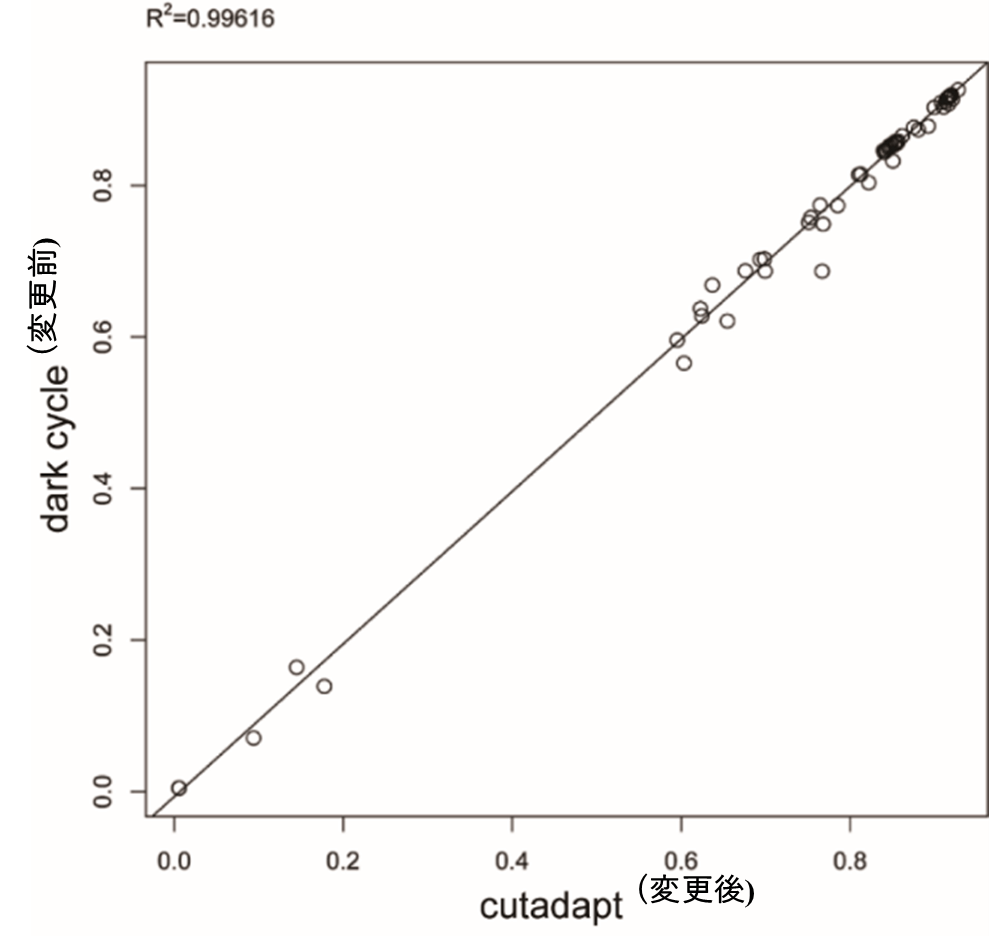
図2. Bray-Curtis距離による統計学的検定
試験に関するお問い合わせ
株式会社テクノスルガ・ラボ 営業部
E-mail:tsl-contact@tecsrg.co.jp
電話番号:054-349-6211
